Bases de datos biológicas¶
Las bases de datos más relevantes en biología incluyen datos de secuencias de nucleótidos, proteínas, estructura de proteínas, genomas, expresión genética, bibliografía, taxonomía, metabolismo, factores de transcripción, etc. Nos podemos hacer una idea de las bases de datos disponibles en esta base de datos de bases de datos biológicas.
Principales bases de datos se secuencias de nucleótidos:
Algunas bases de datos de genomas de organismos concretos:
Principales bases de datos de proteínas:
Bibliografía:
Rutas metabólicas:
Enfermedades genéticas humanas:
Bases de datos de secuencias¶
Una base de datos de nucleótidos es una colección de secuencias de ADN o proteínas.
A estas secuencias normalmente se les añade alguna información relevante y una interfaz para poder acceder a la propia base de datos.
Las tres bases de datos principales de nucleótidos son: EMBL , Genbank y DDBJ.
Intercambian las nuevas secuencias todos los días, por lo tanto, en las tres se encuentra depositada la misma información.
Con el tiempo estas bases de datos han ido añadiendo información adicional a las secuencias y ahora incluyen funciones, mutaciones, proteínas codificadas, referencias bibliográficas, etc.
El crecimiento anual de estas bases de datos es enorme. En el recuento del 15 de Junio de 2007 había más de 73 millones de secuencias en la Genbank y en 2012 mas de 157 millones, y este crecimiento exponencial dista mucho de saturarse.
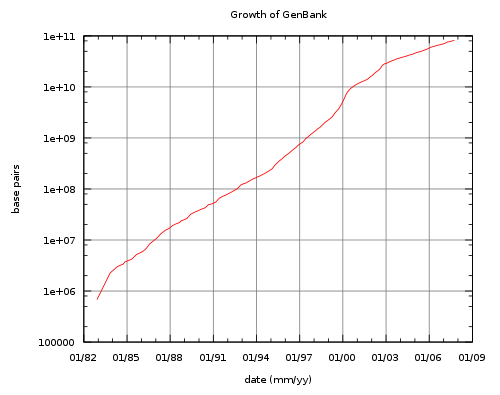
De http://commons.wikimedia.org/wiki/File:Growth_of_Genbank.svg
Dado el gran número de secuencias incluidas las bases de datos suelen estar divididas en secciones para facilitar la búsqueda.
Genbank¶
Genbank es una colección pública de secuencias de nucleótidos anotadas.
Incluye secuencias de ARNm con regiones codificantes, ADN genómico correspondientes a uno o varios genes y ARN ribosómico.
Es un almacén público de secuencias.
No modifican las secuencias sin el consentimiento de los autores.
Es redundante, pueden haber varias secuencias para un gen determinado.
Hay secuencias de muy buena calidad, pero también hay algunas no tan buenas.
Para facilitar la búsqueda han dividido las secuencias en distintas divisiones.
Hay divisiones taxonómicas para las secuencias de alta calidad: primate, rodent, other mamalian, invertebrate, etc.
Otras divisiones se reservan para las secuencias de menos calidad: EST (Expressed Sequence Tag), SRA, GSS (genome survey sequence), HTGS (high throughput genomic sequencing).
En este documento nos resumen las principales características y funcionalidades de la Genbank.
Refseq¶
Refseq (Reference Sequence) es otra base de datos de nucleótidos mantenida por el NCBI.
Es una base de datos secundaria.
Es una colección de secuencias de ADN, ARN y proteínas mantenida y revisada.
Sólo tiene una secuencia por gen y organismo.
Tiene entradas independientes para el ADN genómico, el transcrito y las proteínas.
Sólo incluye los principales organismos (10.854 frente a los más de 160.000 de la Genbank en las versiones de septiembre 2010).
Uniprot¶
Uniprot es una base de datos de proteínas.
Nace por la unión de tres bases de datos previas: Swiss-prot, TrEMBL y PIR-PSD.
Swiss-Prot y TrEML continuan siendo dos secciones en la Uniprot.
Swiss-Prot está compuesta por registros anotados manualmente.
Las proteínas de Swiss-prot tienen una información de alta calidad.
TrEMBL es una traducción automática de las secuencias de la EMBL.
El objetivo es ir anotando manualmente las secuencias de TrEMBL, pero dado el ritmo de secuenciación esta tarea no terminará nunca.
Swiss-Prot es mucho más pequeña que TrEMBL.
PDB, Protein Data Bank¶
PDB alberga información sobre estructura de proteínas y ácidos nucléicos.
Además de la estructura incluye información sobre la secuencia, las condiciones de cristalización, otras proteínas de estructura similar e imágenes 3D.
PubMed¶
PubMed da accesso a la base de datos de referencias bibliográficas MEDLINE.
Su ámbito principal es la medicina, por lo que las revistas de otros campos no siempre están incluidas.
Tiene una buena colección de entradas relacionadas con la bioquímica, biología celular y la medicina.
Incluye los títulos, autores y resúmenes de los artículos publicados.
Muchas de las revistas incluidas permiten el acceso a texto completo del artículo y PubMed permite buscar sólo textos de acceso libre.
Acceso a la información¶
El núcleo de una base de datos está constituido por los datos almacenados de forma estructurada.
Es difícil para un usuario sin una preparación previa acceder a los datos estructurados.
Para simplificar el acceso las bases de datos proporcionan un interfaz fácil de utilizar.
Lo más común es que estos interfaces sean un página web, lo que posibilita que se acceda a los datos utilizando un simple navegador web.
Los interfaces para las bases de datos proporcionan herramientas de búsqueda y ficheros de texto en los que mostrar la información.

El usuario obtiene una serie de ficheros de texto, bien sea en texto plano o en html.
Cada uno de los ficheros incluye la información de uno o varios registros de la base de datos.
Las distintas bases de datos muestras la información en archivos con distintos formatos.
Entrada en formato Genbank
LOCUS EC750390 558 bp mRNA linear EST 03-JUL-2006
DEFINITION POE00005652 PL(light) Polytomella parva cDNA similar to frataxin protein
-related, mRNA sequence.
ACCESSION EC750390
VERSION EC750390.1 GI:110064507
KEYWORDS EST.
SOURCE Polytomella parva
ORGANISM Polytomella parva
Eukaryota; Viridiplantae; Chlorophyta; Chlorophyceae;Chlamydomonadales;
Chlamydomonadaceae; Polytomella.
REFERENCE 1 (bases 1 to 558)
AUTHORS Lee,R.W. and Borza,T.
TITLE The colorless plastid of the green alga Polytomella parva: a repertoire of its functions
JOURNAL Unpublished (2006)
COMMENT Contact: TBestDB
Departement de Biochimie, Universite de Montreal
Montreal, Canada
Email: tbestdb-curator@bch.umontreal.ca
Plate: 4065.
FEATURES Location/Qualifiers
source 1..558
/organism="Polytomella parva"
/mol_type="mRNA"
/db_xref="taxon:51329"
/clone_lib="PL(light)"
ORIGIN
1 gcggccgctt tttttttttt tttttttttt ttttcgtccg ttatttcttt tttaagaatg
61 cagtcatctg tacatcgtca agtattcgga gtgttatctc gttttgtggg aaacaaagcg
121 ggtattttta caaagcataa tcatggtgtc tcaaggttgt cttcatgcac ttcgtcatgc
181 gtaaagatgt atactagcaa caaggccccc gaggatcttc aaacgttcca ccggcaagca
241 gacgaaactc tagagcaagt cactgaagcc cttgaaaact atgtagatga gcatgaagtg
301 gaaggcagcg acattgagca tacgcaagga gtgcttacta ttaagcttgg aactcttgga
361 agttatgtaa ttaataaaca gactcctaat aagcagatat ggttatcctc tcccgtcagt
421 ggacccttcc gatatgatct taaagaaggt gcctgggttt atgaacgggc tggcgaggct
481 cggcgcgagc ttatttctca attagaaaca gaaatttcgg atttagttgg tgtcgaatta
541 aagataagta actgaacg
//
Entrada en formato EMBL
ID EC750390; SV 1; linear; mRNA; EST; PLN; 558 BP.
XX
AC EC750390;
XX
DT 04-JUL-2006 (Rel. 88, Created)
DT 04-JUL-2006 (Rel. 88, Last updated, Version 1)
XX
DE POE00005652 PL(light) Polytomella parva cDNA similar to frataxin
DE protein-related, mRNA sequence.
XX
KW EST.
XX
OS Polytomella parva
OC Eukaryota; Viridiplantae; Chlorophyta; Chlorophyceae; Chlamydomonadales;
OC Chlamydomonadaceae; Polytomella.
XX
RN [1]
RP 1-558
RA Lee R.W., Borza T.;
RT "The colorless plastid of the green alga Polytomella parva: a repertoire of
RT its functions";
RL Unpublished.
XX
DR UNILIB; 42732; 19932.
XX
CC Contact: TBestDB
CC Departement de Biochimie, Universite de Montreal
CC Montreal, Canada
CC Email: tbestdb-curator@bch.umontreal.ca
CC Plate: 4065.
XX
FH Key Location/Qualifiers
FH
FT source 1..558
FT /organism="Polytomella parva"
FT /mol_type="mRNA"
FT /clone_lib="PL(light)"
FT /db_xref="taxon:51329"
FT /db_xref="UNILIB:42732"
XX
SQ Sequence 558 BP; 153 A; 105 C; 127 G; 173 T; 0 other;
gcggccgctt tttttttttt tttttttttt ttttcgtccg ttatttcttt tttaagaatg 60
cagtcatctg tacatcgtca agtattcgga gtgttatctc gttttgtggg aaacaaagcg 120
ggtattttta caaagcataa tcatggtgtc tcaaggttgt cttcatgcac ttcgtcatgc 180
gtaaagatgt atactagcaa caaggccccc gaggatcttc aaacgttcca ccggcaagca 240
gacgaaactc tagagcaagt cactgaagcc cttgaaaact atgtagatga gcatgaagtg 300
gaaggcagcg acattgagca tacgcaagga gtgcttacta ttaagcttgg aactcttgga 360
agttatgtaa ttaataaaca gactcctaat aagcagatat ggttatcctc tcccgtcagt 420
ggacccttcc gatatgatct taaagaaggt gcctgggttt atgaacgggc tggcgaggct 480
cggcgcgagc ttatttctca attagaaaca gaaatttcgg atttagttgg tgtcgaatta 540
aagataagta actgaacg 558
//
Entrada en formato FASTA
>gi|110064507|gb|EC750390.1|EC750390 POE00005652 PL(light) Polytomella parva cDNA
similar to frataxin protein-related, mRNA sequence
GCGGCCGCTTTTTTTTTTTTTTTTTTTTTTTTTTCGTCCGTTATTTCTTTTTTAAGAATGCAGTCATCTG
TACATCGTCAAGTATTCGGAGTGTTATCTCGTTTTGTGGGAAACAAAGCGGGTATTTTTACAAAGCATAA
TCATGGTGTCTCAAGGTTGTCTTCATGCACTTCGTCATGCGTAAAGATGTATACTAGCAACAAGGCCCCC
GAGGATCTTCAAACGTTCCACCGGCAAGCAGACGAAACTCTAGAGCAAGTCACTGAAGCCCTTGAAAACT
ATGTAGATGAGCATGAAGTGGAAGGCAGCGACATTGAGCATACGCAAGGAGTGCTTACTATTAAGCTTGG
AACTCTTGGAAGTTATGTAATTAATAAACAGACTCCTAATAAGCAGATATGGTTATCCTCTCCCGTCAGT
GGACCCTTCCGATATGATCTTAAAGAAGGTGCCTGGGTTTATGAACGGGCTGGCGAGGCTCGGCGCGAGC
TTATTTCTCAATTAGAAACAGAAATTTCGGATTTAGTTGGTGTCGAATTAAAGATAAGTAACTGAACG
Principales campos en el formato Genbank
Más información sobre el formato_en_el_NCBI
Campo |
Descripción |
Búsqueda en Entrez |
|---|---|---|
Locus name |
Un nombre único para la secuencia |
[ACCN] |
Sequence length |
Longitud de la secuencia |
[SLEN] |
Molecule Type |
ADN, genómico, mRNA, etc. |
[PROP] |
Genbank Division |
División a la que pertenece la secuencia |
[PROP] |
Modification Date |
Fecha de la última modificación |
[MDAT] |
Definition |
Breve descrición de la seucuencia |
[TITL] |
Accession |
Identificador único de la entrada, no varía aunuque se modifique la secuencia |
[ACCN] |
Version |
Número de versión de la secuencia |
All fields |
GI |
Identificador único de la secuencia, cambia con las modificaciones |
All fiedls |
Keywords |
Palabras clave que describen la secuencia |
[KYWD] |
Source |
Nombre del organismo |
[ORGN] |
Organism |
Nombre científico del organismo |
[ORGN] |
Reference |
Publicaciones relacionadas |
[TITL] [AUTH] [JOUR] |
Features |
Información sobre las regiones de interés |
[FKEY] |
source |
Longitud de la secuencia, nombre del organismo, taxón ID |
[ALL] |
Taxon |
Taxón ID |
|
CDS |
Secuencia codificante |
[FKEY] |
protein_id |
Identificador de la secuencia protéica |
All fields |
GI |
Identificador único de la secuencia protéica |
All fields |
Translation |
Traducción |
|
gene |
Región cubierta por un gen |
[FKEY] |
El campo features es especialmente importante, describe qué conocimiento se tiene sobre la secuencia.
Búsqueda de ejemplo¶
Podemos buscar en Entrez qué se sabe sobre el síndrome de Usher 1C.
Buscamos: Usher syndrome 1C.
Encontramos:
Pubmed: 22 artículos.
Pubmed Central: 255 artículos.
OMIM: 4 entradas.
Nucleotide: 1138 secuencias.
Protein: 76 secuencias.
Si vamos al Link_OMIM vemos que hay 2 entradas otras enfermedades, una para el síndrome y otra para el gen responsable.
El carácter está localizado en la región cromosómica 11p15.1.
- Desde la pagina del gen de la base de datos OMIME, podemos ebtener informacion sobre el gen en la DB Gene del ncbi:
- El gen (Link_Gene).
Las secuencias de la Ref seq relacionadas.
Podermos ver las publicaciones mas relevantes del gen
Tambien podemos seleccionar las que corresponden a RefSeq (4 sequencias). Entre estas se encuentran dos variantes del mRNA humano (human_b3 y human_1). En los links de las secuencias volvemos a tener la opción de buscar en otras bases de datos de Genes (Gene), Genes homólogos (HomoloGene), bibliografía gratuita (PMC), bibliografía (PubMed), secuencias relacionadas (Related Sequences), etc.
Formatos de secuencia¶
Las secuencias de nucleótidos y proteínas se pueden guardar en distintos formatos. Los más comunes utilizan algún tipo de fichero_de_texto.
Si sólo queremos guardar la secuencia se puede crear un fichero se texto y simplemente escribir la secuencia. Esto se conoce como secuencia en texto plano. El fichero sólo puede contener caracteres IUPAC para secuencia y espacios, los números no están permitidos.
ACAAGATGCCATTGTCCCCCGGCCTCCTGCTGCTGCTGCTCTCCGGGGCCACGGCCACCGCTGCCCTGCCCCTGGAGGGTAC
GGCCCCACCGGCCGAGACAGCGAGCATATGCAGGAAGCGGCAGGAATAAGGAAAAGCAGCCTCCTGACTTTCCTCGCTTGGT
AGTGGACCTCCCAGGCCAGTGCCGGGCCCCTCATAGGAGAGGAAGCTCGGGAGGTGGCCAGGCGGCAGGAAGGCGCACCCCC
ATCCGCGCGCCGGGACAGAATGCCCTGCAGGAACTTCTTCTGGAAGACCTTCTCCTCCTGCAAATAAAA
Secuencia en texto plano.
Este formato tiene algunas limitaciones, no puede haber más que una secuencia dentro del fichero y no puede incluirse el nombre de la secuencia dentro del fichero.
En el formato FASTA la secuencia comienza con un nombre y una descripción. Esta línea se distingue porque siempre comienza con el signo ‘>’. A continuación sigue la secuencia propiamente dicha con el formato en texto plano.
Pueden incluirse varias secuencias en un mismo fichero.
Es un formato muy utilizado.
>nombre_secuencia1 descripción
ACAAGATGCCATTGTCCCCCGGCCTCCTGCTGCTGCTGCTCTCCGGGGCCACGGCCACCGCTGCCCTGCC
CCTGGAGGGTGGCCCCACCGGCCGAGACAGCGAGCATATGCAGGAAGCGGCAGGAATAAGGAAAAGCAGC
CTCCTGACTTTCCTCGCTTGGTGGTTTGAGTGGACCTCCCAGGCCAGTGCCGGGCCCCTCATAGGAGAGG
AAGCTCGGGAGGTGGCCAGGCGGCAGGAAGGCGCACCCCCCCAGCAATCCGCGCGCCGGGACAGAATGCC
CTGCAGGAACTTCTTCTGGAAGACCTTCTCCTCCTGCAAATAAAACCTCACCCATGAATGCTCACGC
>nombre_secuencia2 descripción
ACAAGATGCCATTGTCCCCCGGCCTCCTGCTGCTGCTGCTCTCCGGGGCCACGGCCACCGCTGCCCTGCC
CCTGGAGGGTGGCCCCACCGGCCGAGACAGCGAGCATATGCAGGAAGCGGCAGGA
Secuencia en formato fasta.
El formato fastq se desarrolló para poder almacenar de un modo sencillo la secuencia junto a su calidad. Se utiliza sobre todo para guardar lecturas de secuenciadores de ADN. Se basa en ficheros de texto:
@seq_1
GATTTGGGGTTCAAAGCAGTATCGATCAAATAGTAAATCCATTTGTTCAACTCACAGTTT
+
!''*((((***+))%%%++)(%%%%).1***-+*''))**55CCF>>>>>>CCCCCCC65
@seq_2
ATCGTAGTCTAGTCTATGCTAGTGCGATGCTAGTGCTAGTCGTATGCATGGCTATGTGTG
+
208DA8308AD8SF83FH0SD8F08APFIDJFN34JW830UDS8UFDSADPFIJ3N8DAA
En realidad este formato se inspiró en el fasta. Cada secuencia tiene 4 líneas. En la primera encontramos el nombre tras el símbolo “@” y, opcionalmente, la descripción. La segunda línea tiene la secuencia y la cuarta la calidad codificada como letras.
De este formato existen varias versiones: solexa, Illumina y Sanger, pero la más utilizada es Sanger.
SSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSS......................
..........................XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX......................
...............................IIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIII......................
!"#$%&'()*+,-./0123456789:;<=>?@ABCDEFGHIJKLMNOPQRSTUVWXYZ[\]^_`abcdefghijklmnopqrstuvwxyz{|}~
| | | | | |
33 59 64 73 104 126
S - Sanger Phred+33, raw reads typically (0, 40)
X - Solexa Solexa+64, raw reads typically (-5, 40)
I - Illumina Phred+64, raw reads typically (0, 40)
Pero una secuencia puede tener más atributos además de la propia secuencia y su calidad asociada. Podemos querer incluir el nombre del investigador que la secuenció o un identificador para una base de datos. Los formatos más extendidos con estos atributos extras son el formato_embl y el formato_genbank. También hay formatos que contienen información sobre la calidad de las secuencias como FASTAQ.
Ejemplo de formato Genbank.
Existen otros muchos formatos de secuencia, pero estos son los más comunes. Hay varias utilidades para convertir secuencias de formato a otro, como por ejemplo Readseq.
Ontologías¶
Una ontología, en bioinformática, es un modo de estructurar el conocimiento.
Information Science: the hierarchical structuring of knowledge about things by subcategorizing them according to their essential (or at least relevant and/or cognitive) qualities.
En las bases de datos hay una gran cantidad de información, pero si no se organiza de forma estructurada es difícil tratarla de modo sistemático.
Supongamos que queremos buscar todos los genes implicados en el metabolismo de lípidos, ¿qué buscamos? lipid, lipid metabolism, fat degradation, etc…
Este problema se puede solucionar mediante un vocabulario controlado.
Existen varias ontologías relacionadas con la biología, pero la principal es la Gene Ontology (GO).
El proyecto pretende describir de un modo consistente los genes de distintas bases de datos.
GO se compone de tres ontologías dedicadas a los procesos biológicos, los componentes celulares y las funciones moleculares.
Molecular function: la función que cumple el producto de un gen. Ejemplos: transcription factor, DNA helicase.
Biological process: procesos, como “mitosis” o “metabolismo de purinas”.
Cellular component: estructuras subcelulares, localizaciones, complejos macromoleculares. Ejemplos: nucleo, telomero, origin recognition complex.
Un gen puede tener varios términos GO asociados en cada ontología.
Los términos GO facilitan las búsquedas en las bases de datos y las búsquedas sistemáticas.
Las onotolgías GO están estructuradas.

De http://www.geneontology.org/
Los genes se anotan a un nivel u otro dependiendo de lo que se sepa sobre ellos.
Una ontología se parece a un diccionario, pero se diferencia en un par de aspectos:
Los términos están definidos de forma exacta y no hay sinónimos.
Los términos están ordenados en una estructura jerárquica.
Cada término GO está asociado a un identificador único y una definición concreta.